機械学習の手法などをいろいろ試してみます。
1. UMAP
分析が難しい教師なし学習。そのなかでも強力だと話題のUMAPを試してみます。
パッケージのインストールに苦戦、お手軽とは言えませんが精度などについても検証してみます。
1.1. Install by pip
Ubuntu18.04にインストールしています。
検索してみるとCondaによるインストールする方法が多く紹介されています。
私としてはCondaは使用したくないのでPipによるインストール方法を試してみます。
| PipとCondaを混在させてパッケージ管理していて環境が壊れた経験からCondaを使用しないことにしています。 |
1.1.1. RuntimeError: llvm-config failed executing
手始めにpipでインストールを試してみるとエラーが発生しました。
pip install umap-learnRuntimeError: llvm-config failed executingllvm-configがインストールされていないようです。
1.1.2. llvm-configをインストール
llvm-configのインストールも情報が少なくマニュアルも難しい。
手始めにaptでインストールしてみます。
sudo apt-get install llvmバージョンが古くてエラー
llvm-configをインストール後に再度pipによるインストールを試します。
環境変数”LLVM_CONFIG”を設定してからpipコマンドを実行します。
export LLVM_CONFIG=/usr/bin/llvm-config
pip install umap-learnRuntimeError: Building llvmlite requires LLVM 10.0.x or 9.0.x, got '6.0.0'.1.1.3. llvm-config-9 をインストール
aptコマンドによるインストールではバージョンが古いのでaptコマンドを使用しないで9.0.xをインストールします。
下記ページにしたがってインストールします。
wget https://apt.llvm.org/llvm.sh
chmod +x llvm.sh
sudo ./llvm.sh 9
export LLVM_CONFIG=/usr/bin/llvm-config-9これでllvm-config-9のインストールが完了です。
1.1.4. wheelをインストール
再度pipでumap-learnのインストールを試したところwheelパッケージが存在しないためエラーが発生しました。
pipでインストールします。
pip install wheel1.1.5. umap-learnをインストール
llvm-configとwheelをインストールした結果umap-learnがインストールできました。
pip install umap-learnもしかするとscipyやsklearnが必要なのかもしれません。私の環境ではインストール済みなのでこれらのパッケージインストール作業は省略しています。
1.1.6. インストール作業のまとめ
umap-learnをインストールする作業コマンドをまとめます。
wget https://apt.llvm.org/llvm.sh
chmod +x llvm.sh
sudo ./llvm.sh 9
export LLVM_CONFIG=/usr/bin/llvm-config-9
pip install wheel
pip install umap-learn1.2. umapを試してみる。
クイックに試してみます。
breast_cancerデータを次元削減して2次元の行列を取得しています。
from sklearn.datasets import load_breast_cancer
import umap
X, y = load_breast_cancer(return_X_y=True)
umap_ = umap.UMAP(random_state=0)
embedding = umap_.fit_transform(X)
embeddingarray([[15.124994 , 1.6796212],
[14.847959 , 1.423338 ],
[14.595355 , 3.2353883],
...,
[12.313152 , 9.627517 ],
[14.778064 , 2.3684618],
[-2.9384632, -2.6674404]], dtype=float32)1.3. 精度を確認
UMAPの精度を確認します。
精度はデータによりけりなところがありますが簡略ながら比較してみます。
データはbreast_cancerを使用します。
スコアはf1_scoreを使用します。
1.3.1. ベースライン:lightgbmによる精度
まずは教師あり学習の精度から。
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import cross_val_predict
from sklearn.metrics import f1_score
import lightgbm
X, y = load_breast_cancer(return_X_y=True)
clf = lightgbm.LGBMClassifier()
p = cross_val_predict(
clf,
X,
y,
)
f1_score(y, p)0.9734265734265735スコアは0.97超え。教師あり学習ならほぼ正解出来てしまうデータです。
1.3.2. 比較対象:主成分分析(comp=2)+KMeans
教師なし学習の比較対象として主成分分析を試してみます。
from sklearn.datasets import load_breast_cancer
from sklearn.decomposition import PCA
from sklearn.cluster import KMeans
from sklearn.metrics import f1_score
X, y = load_breast_cancer(return_X_y=True)
kmeans = KMeans(n_clusters=2, random_state=0).fit(X)
f1_score(y, kmeans.labels_)0.8955974842767296想像していたよりも高いスコア。
1.3.3. UMAP(comp=2)+KMeans
今回の目的のUMAP。PCAを超えられるでしょうか?
from sklearn.datasets import load_breast_cancer
import umap
from sklearn.cluster import KMeans
from sklearn.metrics import f1_score
X, y = load_breast_cancer(return_X_y=True)
umap_ = umap.UMAP(random_state=0)
comps = umap_.fit_transform(X)
kmeans = KMeans(n_clusters=2, random_state=0).fit(comps)
f1_score(y, kmeans.labels_)0.8346709470304975…PCAより低いスコアになってしまいました…
PCAよりも高い精度を期待していたのですが想定外です。何かしら検証方法が良くなかったのでしょうか?
このような使用方法にはむいていないのでしょうか?散布図を作成すれば人間にも分かりやすく分離しているのかもしれません。
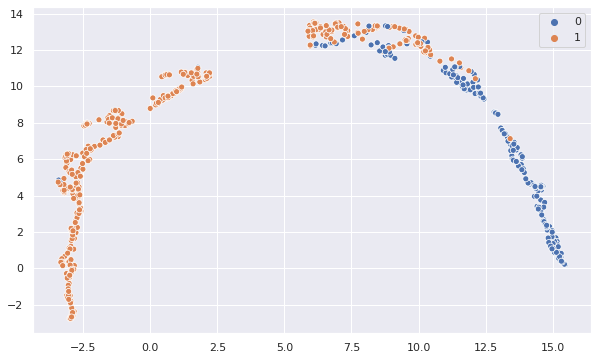
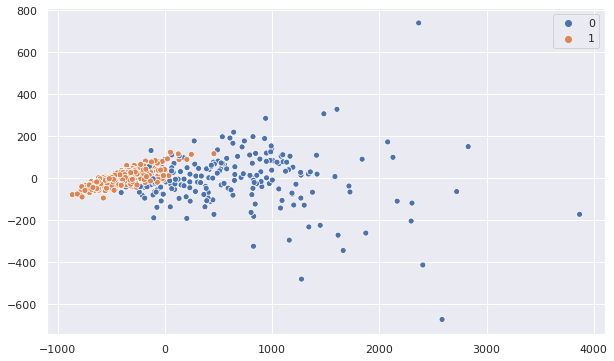
1.3.4. 散布図確認
UMAPとPCAの結果を散布図にしてみます。
両者を比較してみるとUMAPの方が2群がキレイに分離しています。
精度は振るいませんでしたが人間が状況を理解するにはキレイに分離しているUMAPが適していそうです。
2. opencv
画像の処理でopencvを使用することがあります。何度か使用しているのですがインストールなど環境構築方法は都度調べることになってしまっています。良い機会なので環境構築方法について整理しておきます。
2.1. Install
最新バージョンのインストールは簡単にはできなさそうです。
調べてみてこちらの記事を見つけました。記事を公開してくれていることはありがたいことです。
この記事で紹介されているgithub上のスクリプトを使用してインストールしてみます。
git clone https://github.com/milq/milq.git
bash milq/scripts/bash/install-opencv.sh無事にインストールできました。実行時間はかなりかかりました。私のPCが非力なことも原因の1つですが…
あとはpipでpythonのパッケージをインストールします。
pip install opencv-pythonpythonで実行できるか試してみます。
import cv2
cv2.__version__バージョンが表示されて無事実行出来たことが確認できます。
3. 座標と値のリストから行列を作成
実現したいことが言葉でうまく表現できず、解決方法の検索も難しいのだが座標と値のリストから行列を作成したい。
色々と調べた結果、coo_matrixを使用する方法を突き止めた。
座標と値のリストで行列を表現する方法は考えてみればSparseな行列の表現方法だと今にして納得いている。
とにもかくにも実現したいことを上手に表現できないので実例を見てもらいたい。
3.1. サンプルプログラム
import numpy as np
from scipy.sparse import coo_matrix
rows = list(range(4))
cols = list(range(3, -1, -1))
values = [1] * 4
mat = coo_matrix(
(values, (rows, cols)),
shape=(4, 4)
).toarray()
print('rows: ', rows)
print('cols: ', cols)
print('values: ', values)
print('mat: \n', mat)3.2. 実行結果
rows: [0, 1, 2, 3]
cols: [3, 2, 1, 0]
values: [1, 1, 1, 1]
mat:
[[0 0 0 1]
[0 0 1 0]
[0 1 0 0]
[1 0 0 0]]4. 画像配列を新規作成する
マスク画像や背景画像など画像配列を作成したい。
実際にやってみようとしたときに少しだけ実現方法に迷ったのでメモ代わりに残します。
4.1. ホワイト画像を作成する
真っ白だと背景と区別ができないので少しグレーの画像を作成する。
import numpy as np
from PIL import Image
white200 = np.full(
(100, 100, 3),
200,
np.uint8,
)
Image.fromarray(
white200
).save(
'work/white200.jpg'
)4.2. 赤画像を作成する
赤画像を作成する。
-
0値の画像配列を作成
-
Rの値に255を代入
import numpy as np
from PIL import Image
red = np.zeros(
(100, 100, 3),
np.uint8,
)
red[:, :, 0] = 255
Image.fromarray(
red
).save(
'work/red.jpg'
)5. KerasClassifierで画像の2値分類:Binary Classification for images
DeepLearningに苦手意識があり避けてしまう傾向なのでこの辺で慣れておきたい。
データ作成から学習、評価までの一通りの流れを実装します。
sklearnの扱いには慣れているのでsklearnラッパーを使用します。
実装していて気になったことがいくつかある。
DeepLearningを避けているうちにいつのまにかtensolflow.kerasが追加されていた。
今後はtf.kerasが主流になるのだろうか?この記事ではtf.kerasを使用する。
当初は2値分類でsigmoidを使用しようと考えていたが多値分類でsoftmaxのほうが汎用的だと思いなおした。
keras.Sequentialのpredict_probaが将来なくなるというWarningが出る。
でもcross_val_predictの引数method="predict_proba"を指定しないと確率値ではなくクラスが出力されてします。とりあえずいまのところはpredict_probaを指定しておく。将来的に使用できなくなってしまうのだろうか?
5.1. データ作成~学習~評価
正規分布に従う画像データを作成します。
平均が155と145の2種類の画像群を作成してどちらの群に属するかを学習します。
標準偏差は両方とも20とします。
画像サイズは20*20とします。
画像を作成するための関数を用意します。
それぞれの画像を1000個、合計2000個作成します。もろもろの情報を変数として作成します。
そして実際にX, Yを作成します。
モデルを定義します。慣れないうちは構造は使いまわしです。
あとはsklearnの書き方で学習します。クロスバリデーションで精度を評価します。
5.2. コードまとめ
import numpy as np
from tensorflow.keras.models import Sequential
from tensorflow.keras.layers import Dense, Dropout, Flatten
from tensorflow.keras.layers import Conv2D, MaxPooling2D
from tensorflow.keras.wrappers.scikit_learn import KerasClassifier
from sklearn.model_selection import cross_val_predict
from sklearn.metrics import roc_auc_score
rows=20
cols=20
input_shape = (rows, cols, 1)
m_1 = 155
m_0 = 145
sd = 20
n_images_1 = 1000
n_images_0 = 1000
n_images = n_images_1 + n_images_0
def create_x(
m,
s,
rows=20,
cols=20,
):
return np.random.normal(
m,
s,
rows * cols,
).astype(
np.uint8
).reshape(
(rows, cols, 1)
)
X = np.zeros(
(n_images, rows, cols, 1),
dtype=np.uint8
)
for i in range(n_images_1):
X[i] = create_x(m_1, sd)
for i in range(n_images_0):
X[i + n_images_1] = create_x(m_0, sd)
Y = np.zeros(n_images, dtype=np.uint8)
Y[:n_images_1] = 1
def create_model():
model = Sequential()
model.add(
Conv2D(
16,
kernel_size=(3, 3),
activation='relu',
input_shape=input_shape
)
)
model.add(
Conv2D(
32,
(3, 3),
activation='relu'
)
)
model.add(MaxPooling2D(pool_size=(2, 2)))
model.add(Dropout(0.25))
model.add(Flatten())
model.add(Dense(64, activation='relu'))
model.add(Dropout(0.5))
model.add(Dense(2, activation='softmax'))
model.compile(
loss='categorical_crossentropy',
optimizer='adam',
metrics=['accuracy']
)
return model
clf = KerasClassifier(
build_fn=create_model,
epochs=20,
batch_size=10
)
p = cross_val_predict(
clf,
X / 255,
Y,
method='predict_proba'
)
print(roc_auc_score(Y, p[:, 1]))